Neue Einblicke in die Prozessierung von miRNAs
MikroRNAs (miRNAs) sind kleine nicht-kodierende RNAs, die als zelluläre Regulatoren der Genexpression in fast allen biologischen Prozessen eine wichtige Rolle spielen. Sebastian Herzog vom Institut für Entwicklungsimmunologie (Direktor: Andreas Villunger) am Innsbrucker Biozentrum konnte mit seinem Team nun neue, überraschende Details zur Prozessierung dieser miRNAs entlarven. Das Fachjournal Molecular Cell berichtet.
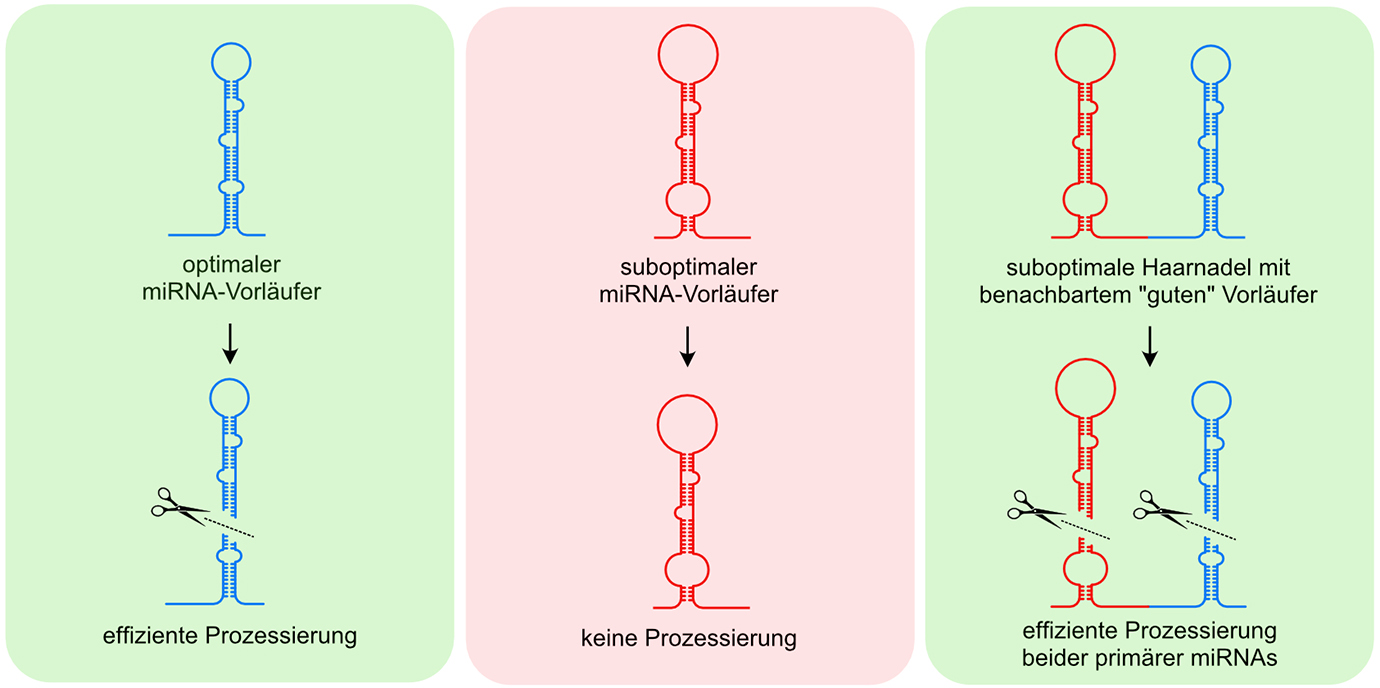
MiRNAs kodieren selbst keine Proteine, beeinflussen aber deren Synthese. Die aus einer 50 bis 80 Nukleotide langen, einzelsträngigen RNA hervorgehenden primären miRNAs verbinden sich in ihren weitgehend komplementären EndBereichen zu einem Doppelstrang und bilden so eine Haarnadelstruktur, die in weiterer Folge von verschiedenen Enzymen bis hin zur reifen miRNA prozessiert wird. Dass diese Haarnadelstrukturen (stem loops) auch suboptimal sein können, fand Sebastian Herzog schon vor mehreren Jahren heraus.
Profitable Nachbarschaft
„Suboptimal bedeutet, dass bestimmte Kriterien wie die Größe des Loops oder die Länge des Stamms nicht den bekannten Regeln entsprechen und die miRNA somit nicht oder nur ineffizient prozessiert werden kann“, erklärt Sebastian Herzog, der im Rahmen eines seiner FWF-Projekte die tumorsuppressive miRNA-15-Familie im Visier hatte, über deren Funktion unter physiologischen und pathophysiologischen Bedingungen kaum etwas bekannt ist.

BU: v.l.: Katharina Hutter, Felix Eichin, Sebastian Herzog, Verena Labi, Michael Lohmüller ©Herzog Lab
In der aus diesem Projekt hervorgegangenen und soeben in Molecular Cell publizierten Arbeit gelang ihm gemeinsam mit seinen beiden PhD-Studierenden Katharina Hutter und Michael Lohmüller und in Kollaboration mit dem Institut für Genomik und RNomik am Biozentrum die Erkenntnis, dass Vorläufer-miRNAs, die nebeneinanderliegen, auch voneinander profitieren. „Wir haben eine ganze Reihe primärer miRNA-Haarnadelstrukturen gefunden, die nicht der Norm entsprechen und daher für sich alleine nicht prozessiert werden. Mithilfe eines zweiten ‚guten‘ Vorläufers auf der gleichen RNA wird dieses Defizit jedoch kompensiert – die suboptimalen profitieren also von den guten in ihrer unmittelbaren Nachbarschaft“, erklärt Herzog.
Unter Anwendung der Genschere CRISPR/Cas9 konnten die Wissenschaftler sogar Gehilfen für diese Art der Prozessierung identifizierten: „In einem groß angelegten Versuch haben wir dazu nach und nach alle Gene in unseren Zielzellen ausgeschaltet und geschaut, wo der Effekt der Nachbarschaftshilfe nicht mehr auftritt. So konnten wir sowohl SAFB2 (scaffold attachment factor B2) als auch ERH (enhancer of rudimentary homolog) Proteine als essentielle Ko-Faktoren für die beschriebene „cluster assistance“ bestimmen“, so Herzog.
Weitere Details dieser Hilfestellung wollen die ForscherInnen in Folgeprojekten aufklären.
(18.06.2020. Text: D. Heidegger, Grafik: Herzog Lab)
Links:
Institut für Entwicklungsimmunologie