Dem Lebenszyklus von Boten-RNA auf der Spur
Forscherinnen und Forscher der Innsbrucker Universitäten haben ein vor drei Jahren entwickeltes Verfahren zur Analyse von Boten-RNA in der Zelle gemeinsam weiterentwickelt. In der Fachzeitschrift Angewandte Chemie International Edition demonstrieren sie, wie mit einem zusätzlichen chemischen Label Produktion und Abbau von Boten-RNA detailliert analysiert werden können.
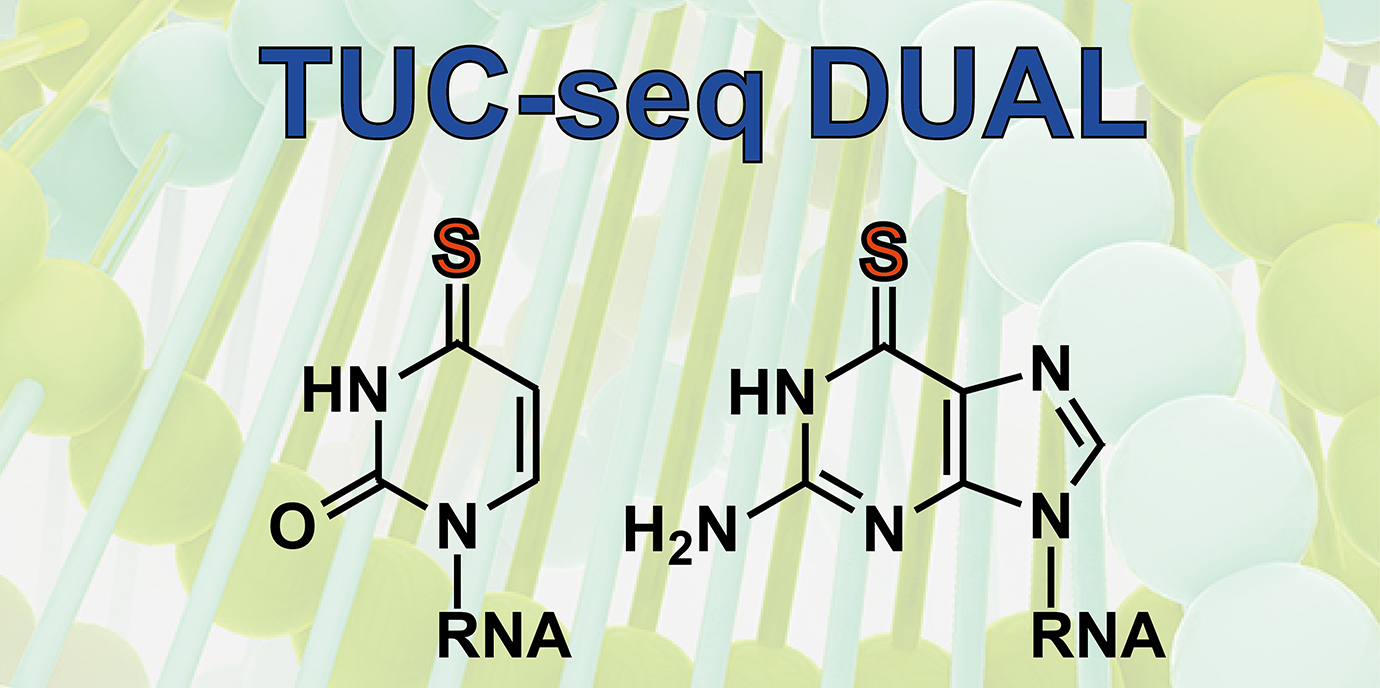
Die genetischen Informationen in der Zelle werden mit Hilfe von Boten-RNA in die Produktionsfabriken für Proteine geliefert. Ihre Funktionsweise ist deshalb für die molekularen Abläufe in der Zelle von großer Bedeutung. Um diese zu verstehen und mögliche Störungen zu erkennen, ist eine detaillierte Analyse dieser Moleküle von großem Interesse. Mit herkömmlichen Sequenzierungsverfahren kann die Menge von Boten-RNA in der Zelle heute rasch und einfach bestimmt werden. „Ob die Konzentration der Boten-RNA aber durch eine gestörte Produktion oder einen fehllaufenden Abbau in der Zelle verändert ist, kann von entscheidender Bedeutung für unser Verständnis von Krankheitsursachen sein“, erklärt die Molekularbiologin Alexandra Lusser vom Biozentrum der Medizinischen Universität Innsbruck. Gemeinsam mit dem Team um Ronald Micura vom Institut für Organische Chemie der Universität Innsbruck und dem Bioinformatiker Dietmar Rieder vom Biozentrum der Medizinischen Universität haben sie ein Verfahren entwickelt, mit dem Entstehung und Abbau von Boten-RNA in der Zelle einfach ermittelt werden können. Die Wissenschaftler füttern dazu die Zellen mit einem Nukleosid, das in die RNA eingebaut wird. Der Schlüssel zum Erfolg liegt in einer chemischen Reaktion, mit der dieses Nukleosid spezifisch in ein anderes umgewandelt wird und dadurch gezielt eine Mutation erzeugt werden kann. Ein Vergleich mit den Sequenzierungsdaten von unbehandelten Zellen gibt Auskunft über die Transkriptions- und Abbaurate der RNA in der Zelle.
Gemeinsamer Erfolg
Nun ist es den Wissenschaftlern gelungen in das Verfahren ein zweites chemisches Label einzuführen und die entsprechende Konvertierungschemie zu entwickeln, mit dem die zeitliche Auflösung dieser Prozesse noch genauer studiert werden kann. „Indem das zweite Label zu einem späteren Zeitpunkt beigemengt wird, können wir in den Sequenzierungsdaten genau bestimmen, welche RNA zwischen der Zugabe des ersten und des zweiten Labels erzeugt wurde, denn nur diese trägt das chemische Produkt des ersten Labels, alle danach produzierten Moleküle zeigen auch die zweite Markierung“, erläutert Ronald Micura den großen Vorteil der neuen Methode. So lässt sich die Quantität der Boten-RNA in einem definierten zeitlichen Fenster sehr gut bestimmen. Damit ist das Verfahren anderen vergleichbaren Methoden deutlich überlegen, ein Erfolg der Wissenschaftlerinnen und Wissenschaftler der Innsbrucker Universitäten, die im Centrum für Chemie und Biomedizin eine gemeinsame Heimat haben. „Wir profitieren sehr von dieser Zusammenarbeit in einer gemeinsamen Forschungsstätte, wo wir Ideen zwischen den Arbeitsgruppen austauschen und neue Ansätze rasch ausprobieren können, unabhängig von der institutionellen Anbindung“, sagt Alexandra Lusser.
Das gemeinsam entwickelte Verfahren wurde inzwischen zum Patent angemeldet und stößt auf reges Interesse der Biotech-Industrie. Die Forschungen in Innsbruck wurden unter anderem vom österreichischen Wissenschaftsfonds FWF und der österreichischen Forschungsförderungsgesellschaft FFG finanziell unterstützt.
(10.03.2020; Text: C. Flatz / Bild: Micura, Lusser)
Links:
Institut für Organische Chemie, Universität Innsbruck
Centrum für Molekulare Biowissenschaften (CMBI), Universität Innsbruck
Sektion für Molekularbiologie, Medizinische Universität Innsbruck
Sektion für Bioinformatik, Medizinische Universität Innsbruck