Innsbrucker Forscher liefern wichtigen Beitrag zur Entschlüsselung des Vaccinia Virus
Eine im Journal CELL veröffentlichte Arbeit gibt erstmals Einblicke in die molekularen Grundlagen des RNA Polymerase Komplexes des Vaccinia Virus. Dieses DNA-Virus bildet die Basis für Pockenschutzimpfstoffe. Den Innsbrucker Wissenschaftern Alexander Hüttenhofer, Direktor des Instituts für Genomik und RNomik, und Forschungsgruppenleiter Matthias Erlacher ist es im Rahmen einer internationalen Forschungsarbeit gelungen, ein Schlüsselmolekül im RNA Polymerase Komplex zu identifizieren, eine tRNA.
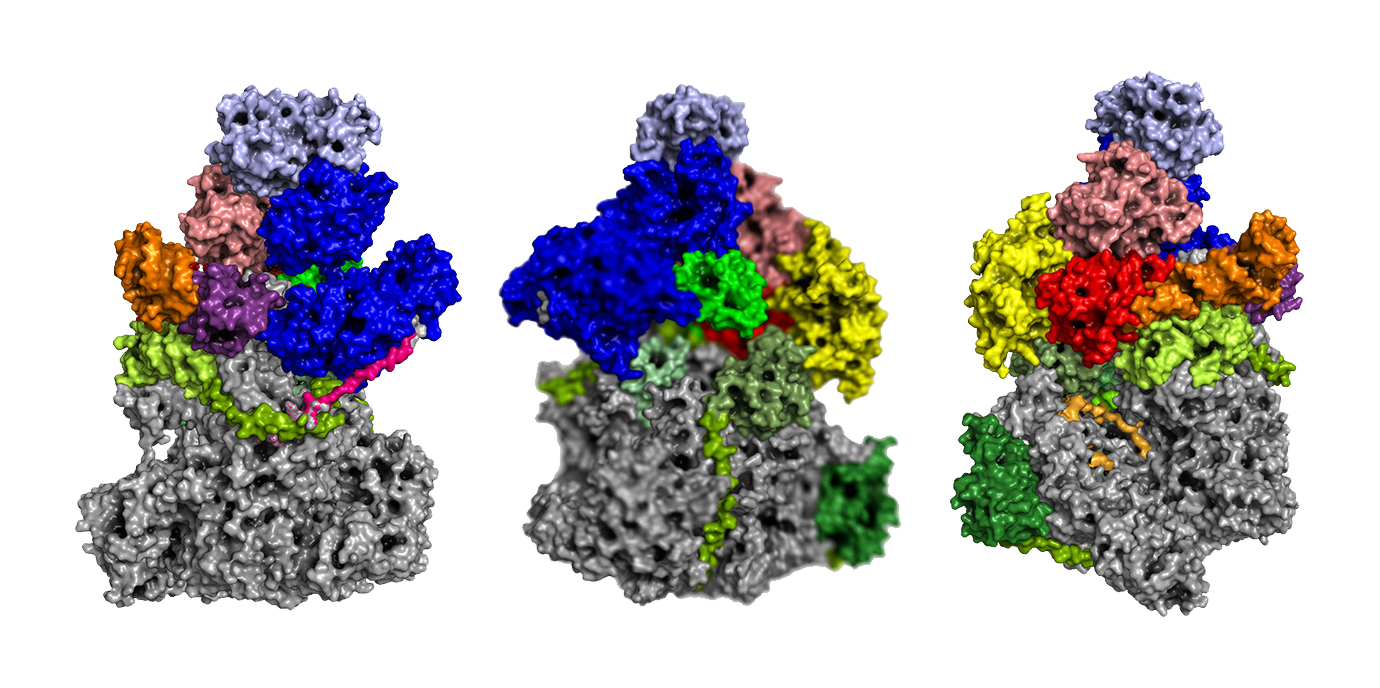
Die Forschungsarbeit von den beiden ErstautorInnen Clemens Grimm aus Würzburg und Hauke S. Hillen aus Göttingen gibt erstmals völlig neue Einblicke in die molekulare Struktur des Vaccinia Virus. Dieses DNA-Virus zählt zu den Pockenviren und ist für die Wissenschaft von besonderem Interesse, da es sich, anders als andere DNA-Viren, nicht im Zellkern des Wirts repliziert, sondern im Cytoplasma. Es generiert dabei seine eigene RNA-Polymerase, welche die genetische Erbinformation des Virus, die auf DNA Ebene gespeichert ist, in RNA (Ribonukleinsäure) übersetzt Diese RNA liefert dann die Erbinformation an Ribosomen, welche daraus virale Proteine generieren. Für das Verständnis der molekularen Grundlagen des Vaccinia Virus ist die RNA-Polymerase Struktur daher essentiell und dementsprechend ist die Erstbeschreibung des komplexen Proteinsystems der RNA-Polymerase ein wichtiger Beitrag zum besseren Verständnis der Funktionsweise des DNA-Virus und möglichen therapeutischen Interventionen. Die beiden Innsbrucker Wissenschafter Alexander Hüttenhofer und Matthias Erlacher von der Medizinischen Universität Innsbruck konnten dabei einen wichtigen Beitrag leisten, da es etwas ihnen gelungen ist, woran zuvor einige Firmen gescheitert waren: Die Identifizierung einer nicht-Protein-kodierenden RNA (ncRNA) innerhalb des RNA-Polymerase Komplexes, welche zwar als RNA zu erkennen war, allerdings nicht auf Sequenzebene beschrieben werden konnte.
Hohe Expertise am Standort: Sensormolekül konnte sequenziert werden
„Wir sind aufgrund unserer ausgewiesenen RNA Expertise am Institut für Genomik und RNomik von den Kollegen aus Würzburg kontaktiert worden, da es anderen Gruppen und Firmen zuvor nicht gelungen war, diese RNA zu sequenzieren“, sagt Alexander Hüttenhofer. „Wir haben uns dann eine andere Strategie überlegt, und die RNA zunächst in Stücke zerlegt, diese Teile sequenziert und dann bioinformatisch wieder zusammengefügt.“ Die Forschungsarbeit unterstreicht dementsprechend auch die hohe Expertise an der „Deep Sequencing Core Facility“ im Bereich der RNA Sequenzierung, deren Leiter Alexander Hüttenhofer vom Institut für Genomik und RNomik am Biozentrum in Innsbruck ist.

BU: Matthias Erlacher (li.) und Alexander Hüttenhofer sind Experten im Bereich der RNA Sequenzierung. (c) Andreas Friedle
Das Ergebnis war dann aber auch für die beiden RNA-Experten Hüttenhofer und Erlacher durchaus überraschend, denn sie haben damit ein Schlüsselmolekül innerhalb der viralen RNA-Polymerase sequenziert und damit einen wesentlichen Beitrag zur Erstbeschreibung dessen Mechanismus geliefert. „Wir konnten zeigen, dass es sich um eine Glutamin tRNA handelt, wobei der Virus Glutamin als Energiequelle nutzt und nicht wie sonst üblich Glukose. Die sogenannte Transfer-RNA dient dabei normalerweise als Hilfsmolekül bei der Proteinbiosynthese“, ergänzt Matthias Erlacher. „Wir haben hier aber ein RNA Sensormolekül mit einer neuen Funktion innerhalb des RNA Polymerase Komplexes des Virus entdeckt, dass der Virus vermutlich benötigt, um festzustellen ob es noch genügend Energie in der Wirtzelle gibt, oder ob es Zeit ist die Zelle zu verlassen.“ In einer weiteren Forschungsarbeit, zusammen mit der Arbeitsgruppe von Prof. Utz Fischer in Würzburg, soll nun künftig die Funktion der Glutamin tRNA untersucht werden. Für das genaue Verständnis des Vaccinia Virus ist es dabei notwendig, die molekularen Strukturen detailliert zu entschlüsseln: nur wenn die molekularen Grundlagen genau bekannt sind, wird es zukünftig auch möglich sein, das Virus und insbesondere seine pathogenen Verwandten entsprechend therapeutisch zu beeinflussen erklärt Alexander Hüttenhofer.
(7.1.2020, B. Hoffmann-Ammann)
Links:
Structural Basis of Poxvirus Transcription: Vaccinia RNA Polymerase Complexes, Cell (2019)
AutorInnen: Clemens Grimm, Hauke S. Hillen, Kristina Bedenk, Simon Neyer, Qian Zhang, Alexander Hüttenhofer, Matthias Erlacher, Christian Dienemann, Andreas Schlosser, Henning Urlaub, Bettina Böttcher, Aladar A. Szalay, Patrick Cramer und Utz Fischer.