Neue Erkenntnisse zu grundlegendem Zellmechanismus
Ein Forschungsteam der Medizinischen Universität Innsbruck hat erneut mit einer fundamentalen Entdeckung auf dem Gebiet der RNA (Ribonukleinsäure) für Aufsehen gesorgt: Die Untersuchungen unter der Federführung von Matthias Erlacher und Thomas Hoernes von der Sektion für Genomik und RNomik (Direktor: A. Hüttenhofer) geben neue Einblicke in die Proteinsynthese und sind erst kürzlich im renommierten Forschungsjournal „Nature Communications“ veröffentlicht worden.
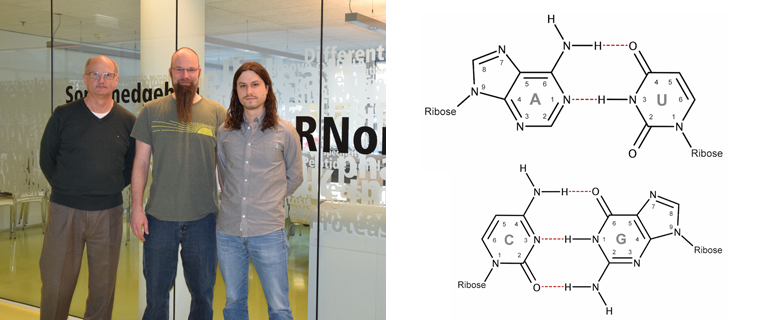
An der Sektion für Genomik und RNomik beschäftigen sich die ForscherInnen mit den grundlegenden Prozessen des menschlichen Lebens. Neue Erkenntnisse verbessern das Verständnis über die genetischen Grundlagen unserer Welt. Die Translation von mRNAs (Messenger RNA) in Proteine ist ein zentraler Prozess in jeder Zelle. Matthias Erlacher und seinen KollegInnen versuchen mit neuen Ansätzen und Techniken, diesen Prozess genauer zu beleuchten. In der Zelle wird die genetische Information für Proteine zuerst in RNA überschrieben und dann von den Ribosomen in eine Abfolge von Aminosäuren übersetzt. Die Proteinbiosynthese basiert auf der korrekten Interaktion von mRNA Codons mit dem Anticodon der jeweiligen tRNA (Transfer RNA), welche die gerade einzubauende Aminosäure trägt. In ihrer Forschungsarbeit untersuchten Erstautor Thomas Hoernes und Letztautor Matthias Erlacher die Innsbrucker diesen Dekodierungsprozess auf atomarer Ebene. „Wir konnten zeigen, dass für die Stabilität dieser Interaktion am Ribosom nicht die Anzahl der Wasserstoffbrücken verantwortlich ist, sondern überwiegend die Geometrie“, erklärt Erlacher. Für die Studie haben die Innsbrucker Forscher unter anderem die Stabilität des Inosin-Cytosin-Basenpaars angeschaut. „Wir wollten wissen, welchen Einfluss weniger stabile Basenpaare auf die Proteinbiosynthese haben “, erklärt Erlacher. „Es hat sich gezeigt, dass das Vorkommen einzelner Inosine in der mRNA kein Problem darstellt, mehrere Inosine die Translation aber inhibieren.“
Erkenntnis spielt Rolle für Serotoninrezeptor im Gehirn
Die Arbeit ist auch deshalb so spannend für die Wissenschaft, da Inosine auch in menschlichen mRNA vorkommen. In der mRNA des Serotoninrezeptors HT2c sind gleich 5 Inosine in unmittelbarer Nachbarschaft zu finden. „Es stellt sich daher die Frage, ob durch diese Konstellation auch die Translation dieser mRNA in neuronalen Zellen betroffen ist“, meint Erlacher. Bereits am Anfang des Jahres war es dem Forschungsgruppenleiter gemeinsam mit dem PhD-Studierenden Hoernes gelungen, zu zeigen, wie Zellen bei der Proteinherstellung einen Punkt setzen. https://www.i-med.ac.at/mypoint/news/714654.html
Die neuerliche Publikation in dem anerkannten Fachjournal Nature Communication bestätigt die hohe Expertise der Life Science in Innsbruck. „Ein wichtiger Erfolgsfaktor ist für uns die interdisziplinäre Zusammenarbeit – nur vereinte Kräfte ermöglichen grundlegende Erkenntnisse“, meint Erlacher. An dieser Forschungsarbeit, die vom FWF/Matching Funds gefördert worden ist [P 30486-BBL und 28494-BBL], waren ForscherInnen der Core Facility Proteinanalytik (Leiter: Herbert Lindner) der Medizinischen Universität Innsbruck sowie aus dem Bereich der Organischen Chemie der Leopold-Franzens-Universität Innsbruck beteiligt. Aus Strassburg hat der renommierte RNA Forscher Eric Westhof mitgearbeitet.
(B. Hoffmann-Ammann)
Publikation:
Translation of non-standard codon nucleotides reveals minimal requirements for codon-anticodon interactions (Nature Communicationsvolume 9, Article number: 4865 (2018))
AutorInnen: Thomas Philipp Hoernes, Klaus Faserl, Michael Andreas Juen, Johannes Kremser, Catherina Gasser, Elisabeth Fuchs, Xinying Shi, Aaron Siewert, Herbert Lindner, Christoph Kreutz, Ronald Micura, Simpson Joseph, Claudia Höbartner, Eric Westhof, Alexander Hüttenhofer & Matthias David Erlacher
Weitere Information:
Sektion für Genomik und RNomik